现代计算机主要由顺序计算的硅基场效应晶体管组成,即每一个指令集均通过处理器一直在不断地执行程序计数器指向的指令,再更新程序计数器,使其指向下一条指令。而兴起于上世纪90年代的DNA分子计算基于的是平行计算的原理。若把一对DNA分子互补碱基的计算能力理解为1的话,1uM的DNA序列可以平行计算大约1017的数据量。借助于DNA超大的数据平行计算容量,理论上DNA计算可以在一秒内完成比现有的超级计算机更多的操作。同时,DNA分子具有高密度信息储存的能力,1g DNA可以在1cm3的体积中储存约750TB的信息。因此,若结合DNA的平行计算能力和分子识别能力,则可实现精细、智能和复杂的分子计算,在需要借助多靶标多参数的复杂分子分型和疾病诊断中有着较大的应用潜力,是当前智能科学领域的研究热点之一。然而,受限于生物样品中靶标物(DNA/RNA)的浓度过低,且背景环境复杂,DNA计算很少进入到临床疾病的实际诊断中。
2020年5月25日,Nature Nanotechnology(自然·纳米技术)杂志以“Cancer diagnosis with DNA molecular computation”为题在线发表了上海交通大学韩达课题组关于DNA分子计算的最新研究成果,该研究将机器学习算法模型通过DNA计算在分子水平上实现。该DNA计算方法可原位同时分析血清样本中多个miRNA的表达谱,在不需要人工干预和复杂仪器的情况下快速给出肺癌诊断结果,成功将分子计算应用到临床诊断中,为肿瘤的无创分子诊断提供了新途径。
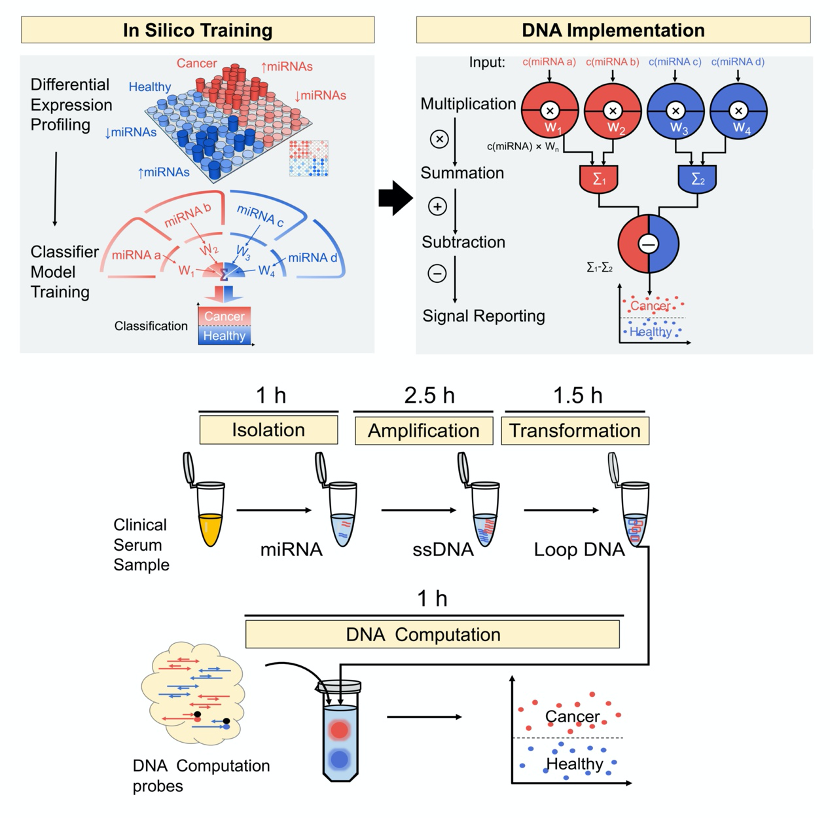
研究人员首先利用TCGA数据库中肺癌miRNA表达谱进行机器学习模型训练,通过对数据库中915例非小细胞肺癌和105例健康人的miRNA表达谱进行差异表达分析和支持向量机(SVM)训练,获得了一组非小细胞肺癌高度相关的特征miRNA及它们各自对应权重的分类模型。该模型训练集准确率达到98.7%,验证集准确率达到98.3%,在理论计算层面实现精准非小细胞肺癌诊断。
随后作者们设计了miRNA的原位信号放大与信号转换方法,将血清中微量的特征miRNA通过线性扩增技术稳定等比例放大到nM数量级。之后通过设计成环反应将只有22nt的miRNA序列转换成长链环状DNA,来满足DNA计算所需的序列设计空间。最后通过DNA计算来实现SVM模型中的乘法、加法和减法,在分子水平实现分类模型的计算公式,使得只有肺癌或健康一种特征的信号分子被放大并产生报告信号。在30例合成样本的验证中,该方法灵敏度和特异性分别达到100%和93.3%,准确率达到96.7%,22例临床实际样本检测中灵敏度和特异性分别达到92.9%和86.7%,准确率达到86.7%。
该研究成功将DNA计算技术应用于解决临床疾病诊断中,其快速、低成本、低误差以及不依赖传统诊断方法的人工数据分析要求等优势,预期可以激发更多的临床应用,实现无创早期肿瘤原位诊断及复发监测。同时该方法也为DNA分子计算在生物医学领域的应用提供了新的启发。
论文链接:https://doi.org/10.1038/s41565-020-0699-0




